 Au moment où beaucoup d’entre nous se demandent comment être utiles pour aider les scientifiques à comprendre la maladie, puis à mettre au point des traitements et in fine un vaccin, Lonni Besançon nous propose de participer à un projet collectif en mettant à leur disposition une partie des ressources matérielles de nos ordinateurs. Pascal Guitton
Au moment où beaucoup d’entre nous se demandent comment être utiles pour aider les scientifiques à comprendre la maladie, puis à mettre au point des traitements et in fine un vaccin, Lonni Besançon nous propose de participer à un projet collectif en mettant à leur disposition une partie des ressources matérielles de nos ordinateurs. Pascal GuittonSi durant vos heures de confinement vous passez quelques minutes sur les réseaux sociaux ou devant les médias, vous avez sûrement entendu parler des raisons pour lesquelles le confinement a été mis en place. C’est l’une des solutions pour espérer réduire l’impact du virus. Parmi les espoirs des scientifiques et du reste de la population pour stopper la propagation du virus reste le développement d’un vaccin ou d’un traitement efficace. Que vous ayez une formation scientifique ou non, il s’avère que chacun peut apporter sa pierre à l’édifice dans la compréhension du virus via l’installation d’un simple programme sur son ordinateur. Ce programme, Folding@Home, repose sur la notion de calcul distribué validée initialement par le projet SETI@Home.

Folding@Home est donc un projet de calcul distribué qui permet de simuler la dynamique des protéines, y compris leur processus de repliement et leurs mouvements quand elles sont impliquées dans diverses maladies. Ces simulations demandent une énorme puissance de calcul que les scientifiques ne peuvent obtenir sur leurs seuls ordinateurs. L’idée de Folding@Home est donc d’utiliser les processeurs et cartes graphiques d’autres personnes, qu’elle soient chercheur.e.s ou non, afin de permettre de créer ces simulations plus rapidement. La liste des maladies étudiées par le projet Folding@home inclut Alzheimer, Parkinson ou Huttington et depuis peu COVID19. Le résultat de ses simulations permets aux scientifiques de mieux comprendre des mécanismes biologiques et donc de créer de nouvelles thérapies.
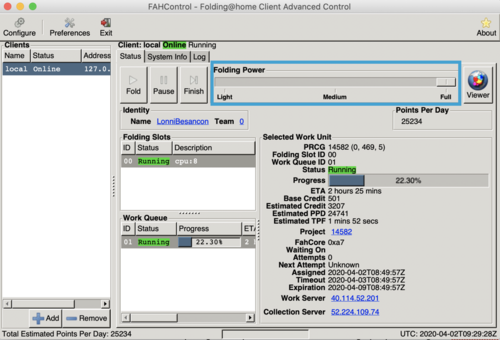
Image 1 : l’écran d’accueil de Folding@Home
Comment participer à cette formidable initiative ? C’est très simple. Il suffit d’abord de télécharger le programme via la page de téléchargement de Folding@Home puis de l’installer sur votre ordinateur. Il suffit ensuite de lancer FAHControl afin de commencer à partager son processeur et/ou sa carte graphique. Ce logiciel permet de contrôler les ressources que l’on donne au projet. Par exemple, le slider accessible sous “Folding Power” permet de déterminer la quantité de puissance de calcul que l’on souhaite donner au projet (voir l’Image1). Le logiciel permet aussi de s’identifier avec un nom d’utilisateur (via le bouton “Configure” et dans l’onglet “Identity”) afin de voir et comparer ses statistiques par rapport à d’autres utilisateurs.
Le programme Folding@Home est configuré pour automatiquement utiliser le processeur et la carte graphique (si elle est compatible, ce qui est rarement le cas sur les ordinateurs portable de chez Apple par exemple) de chaque ordinateur. Le client Folding@Home télécharge d’abord une unité de travail pour une protéine spécifique, commence un calcul sur cette unité de travail et, une fois le calcul achevé, envoie les résultats directement au projet avant de télécharger une nouvelle unité de travail. L’initiative Folding@Home est donc un moyen simple pour chaque citoyen possédant un ordinateur relativement récent et une puissance de calcul assez importante de contribuer à des projets scientifiques.
Lonni Besançon (Linköping University)




Répondre à cet article
Suivre les commentaires : |
|
